1.前言
nnUnet原代码是在Linux下运行,如果现在windows下安装的话,会报各种错误,得改很多的地方,所以可以直接下载nnUnet_windows文件,这是我已经在windows上成功编译的版本,只要安装成功就可以直接运行。该链接中还包含apex安装包以及制作自己数据格式的代码。
nnUnet的版本是2021/7月更新的。
2.安装虚拟环境与Pytorch
最好要安装虚拟环境,否则要是把python环境安装坏了,那就麻烦了。
1.在命令行输入一下代码,其中nnUnet是虚拟环境名称,选择的python3.7版本
conda create -n nnUnet python=3.7
2.激活虚拟环境输入
activate nnUnet
如果退出虚拟环境使用命令deactivate
3.安装Pytorch,我选的是最新的版本pytorch1.9.0,cuda版本为10.2,这个就不详细叙说了,网上有很多教程。
3.安装apex
在上面给的链接中 有apex安装包,或者你可以在官网上下载apex
然后cd 到apex文件下目录,输入命令
pip install -v --no-cache-dir --global-option="–cpp_ext" --global-option="–cuda_ext" ./
如果这个命令报错的话就换下面命令试试
python setup.py install

这里我盗用一下网上的截图,因为我忘了截图了,如果是success则表示安装成功。有别的教程说要按照hiddenlayer,我还没有安装,反正没有出现问题,出现问题再说。如果想安装的先安装git,conda install git。然后以下输入命令
pip install --upgrade git+https://github.com/nanohanno/hiddenlayer.git@bugfix/get_trace_graph#egg=hiddenlayer
4.安装nnUnet
你可以下载我上面提供的代码,你也可以下载nnUnet官方代码,但是在windows下需要改很多地方。
cd 到nnUnet文件目录,输入命令,别丢了一个".",否则你无法安装。
pip install -e .
如果没有报错的话,恭喜你安装成功,如果报错的话,建议你在官网下载最新的代码,然后升级到最新的python库以及pytorch版本。
5.运行nnUnet
如果你是第一次使用nnUnet的话,建议你先下载官方数据集跑一遍,如果成功的话,在制作自己的数据集。
1.创建文件夹
在nnUnet文件目录下创建Dataset文件夹,当然你也可以在其他地方创建其他名字。

下面创建的文件夹一定要一样,在Dataset文件夹下创建三分文件夹,如下图所示,其中nnUnet_preprocessed用于预处理后的数据,nnUnet_raw用于存储原数据和裁剪的数据,nnUnet_trained_models用于存储训练模型与日志。

然后在nnUnet_raw文件夹下载创建如下两个文件夹,nnUnet_cropped_data用于存放预处理数据, nnUNet_raw_data用于存放原数据。

- 设置文件路径,我从官网下载的是Task08_HepaticVessel数据集,随便找一个地方解压。
关键地方来了,这是所有教程没有提到的地方
在linux下要将上面创建的文件添加到系统环境中,但是在windows下没法操作,所以在nnUnet文件夹下找到paths.py文件,然后修改一下三处,注释掉红色框出来的地方,然后换成黄色框出来的地方,文件目录对应你自己的路径。注意:文件路径千万不要写成“//”,就像我下面写的那样就行。否则你会遇到各种错误,这就是windows一直报错的最大原因。

3.转换数据格式,使用以下命令
nnUNet_convert_decathlon_task -i D:\Task08_HepaticVessel\Task08_HepaticVessel
上面是我的路径,你可以换成自己的路径,但我不建议在命令行中运行代码,否则报错的话就很难查到原因,所以可以用下面的方式运行。下面又是干货。
在nnunet\experiment_planning文件夹下找到nnUNet_convert_decathlon_task.py,打开pycharm,然后点击图中地方


在红色框中输入-i D:\Task08_HepaticVessel\Task08_HepaticVessel,然后运行即可,这样报错的话,你可以慢慢调试,寻找错误的地方。
如果运行成功的话,你会在nnUNet_raw_data文件夹下得到如下文件与数据。imagesTr存放的是训练数据,imagesTs是存放测试数据,labelsTr存放的是训练数据的标签, dataset.json是对数据的说明,和存放数据地址。

4.对数据进行预处理,同样不建议使用命令行代码运行,在相同目录下找到nnUNet_plan_and_preprocess.py文件,输入参数
-t 8
其中8对应数据的Id,如果是下载我编译好的代码的话,可以直接运行,如果下载的官方的代码,你会在这遇到一堆问题。
当然你可能还会遇到一个问题,就是电脑cpu内存不够,我电脑的配置是32g内存依然不够,所以我在裁剪数据的时候,只挑选了其中一部分数据集。
如果运行没报错的话,你会在nnUnet_preprocessed文件夹下生成如下的文件夹以及数据

5.开始训练
如果前面一切顺利的话,后面就可以直接运行了在命令行中输入
nnUNet_train 3d_fullres nnUNetTrainerV2 8 4,
如果你想调试的话,但是你会发现找不到nnUNet_train文件,这是集成好的nnUNet_train.exe,位置在你安装虚拟环境中。所以换一个文件运行。
在文件夹nnunet\run下找到run_training.py,输入参数,3d_fullres nnUNetTrainerV2 8 4。其中8代表你的任务ID,4代表五折交叉验证(0代表一折)。
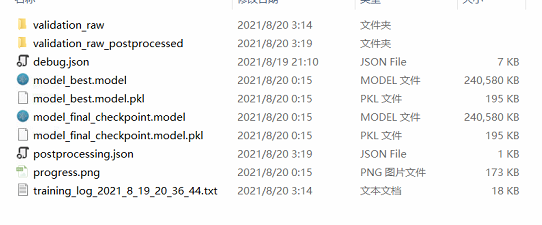
运行成功后你会在如下文件路径中得到训练的日志以及模型。如果少一个文件的话都有可能代表你运行失败,会影响后面的推理。
6.推理
在windows下使用多线程用的是
训练完成后,找到imagesTs路径,就是上面制作数据集的路径,然后输入命令pathos库,所以可能需要安装,输入命令
pip install pathos
然后推理的时候输入命令
nnUNet_predict -i F:\nnUNet_windows\DataSet\nnUnet_raw\nnUNet_raw_data\Task008_HepaticVessel\imagesTs -o F:\nnUNet_windows\DataSet\nnUnet_raw\nnUNet_raw_data\Task008_HepaticVessel\imagesTs_infer -t 8 -m 3d_fullres -f 4
其中-i 参数的是输入路径,-o 参数是输出路径。
如果你想调试的话,在inference文件夹中找到predict_simple.py文件,然后输入参数就可以调试了。

近期评论